유전자 발현 조절 핵심 인자인
‘RNA 결합단백질’ 기작 규명 기대
"Chemical RNA digestion enables robust RNA-binding site mapping at single amino acid-resolution"
RNA와 결합하는 ‘RNA 결합단백질(RNA-binding protein)1)’상에서 정확한 결합 부위를 알 수 있게 됐다.
기초과학연구원(IBS, 원장 노도영) RNA 연구단 김빛내리 단장(서울대 석좌교수)·김종서 연구위원(서울대 책임연구원)은 사람 세포 속 RNA 결합단백질 상에서 결합을 형성하는 ‘RNA 결합자리(RNA-binding site)2)’를 광범위하고 정확하게 찾아낼 수 있는 기법을 개발했다. RNA 결합단백질은 유전자 발현을 조절하는 핵심 인자로, 질병과 세포 기능에 관련된 단백질의 조절 기작을 밝히는 데 기여할 것으로 기대된다.
RNA는 DNA로부터 각 단백질에 해당하는 정보가 전사된 유전체다.
RNA는 이 정보를 번역해 단백질을 생산하는데, 전사되고 나서도 번역 효율, 안정성, 세포 내 위치 등 단백질 생산과정이 조절될 수 있다. 이러한 전사 후 조절은 RNA 결합단백질이 RNA에 붙으면서 이뤄진다.
대부분의 단백질은 전사 후 조절을 거치면서 기능을 가지는데, 핵심 인자인 RNA 결합단백질과 RNA 사이 결합 원리와 상호작용은 거의 밝혀지지 않았다. 복잡한 단백질 구조에서 어느 조각이 결합자리인지조차 정확히 알 수 없었기 때문이다.
RNA 결합자리를 보기 위해서는 작은 단백질 조각의 질량을 측정해, 해당 조각을 구성하는 아미노산 및 단백질 내 위치를 추론하는 질량분석(mass spectrometry)3) 방법을 쓴다.
RNA 결합단백질-RNA 결합체를 효소로 잘게 쪼개면 단백질 조각인 펩타이드(peptide)4)에 RNA 조각이 붙은 형태가 된다. 이 질량 구성을 측정하고 RNA가 붙지 않은 펩타이드와 비교하면, RNA가 붙은 아미노산 자리는 그 질량만큼 차이가 나게 된다.
그러나 기존 연구에서는 RNA가 완전히 분해되지 않고 남아있는 RNA 조각 크기가 제각각이어서 질량 측정에 이 오차를 고려해야 했다. 오차를 고려하고도 확실하게 RNA 조각이 붙었다고 판단되는 아미노산 자리만 알 수 있었다는 것이다. 따라서 1천 개 이상 RNA 결합단백질에서 한 번에 수십~수백 개 RNA 결합자리만 확인 가능했으며, 위치의 정확도도 떨어졌다.
연구진은 기존에 쓰이던 효소 대신 불산을 이용해 문제를 해결했다. 불산은 RNA를 동일한 분자 한 개로 완전히 분해해, 한 번에 2,000개 RNA 결합자리를 찾아낼 수 있었다.
불산 처리 후 RNA 조각의 질량을 쟀더니 동일한 유리딘(Uridine) 5) 분자만 남음을 확인했다. RNA 조각의 질량 오차를 줄임으로써 RNA 결합자리를 더 많이 알아낼 수 있었던 것이다. 결과적으로 세포 전체 RNA에 결합한 600 개의 RNA 결합단백질 내에서 약 2,000 종류 RNA 결합자리를 아미노산 수준의 고해상도로 찾아낼 수 있었다.
이렇게 찾은 RNA 결합자리를 바탕으로 연구진은 새로운 가설들을 제시했다. 먼저 질병 및 세포 기능에 중요한 단백질에서 RNA 결합자리를 다수 발견했다.
일례로 근위축성 측삭 경화증의 원인 단백질인 TDP-43, DNA 복구에 필수적인 PRKDC에 존재하는 RNA 결합자리를 찾았다. 이는 기존에 알려지지 않았던 RNA와의 결합이 각각 단백질의 기능을 조절할 수 있음을 시사한다. 또 RNA 결합자리가 단백질의 공유결합 변형 자리와 비슷함도 보였다. RNA 결합자리의 결합력 조절 원리가 공유결합 변형일 수 있다는 뜻이다.
이번에 규명한 RNA 결합자리를 토대로 세포 내 RNA-RNA 결합단백질 상호작용을 세밀하게 연구할 수 있을 것으로 보인다. 또한 이번 기술을 변형해 RNA 뿐만 아니라 DNA와 결합하는 단백질로도 확장할 수 있으리라 기대된다.
이번 연구성과는 Nature Structural & Molecular Biology(IF 12.109)에 6월 9일 0시(한국시간) 온라인 게재됐다.
[참조]
1) RNA 결합단백질(RNA-binding protein) : RNA 서열 또는 구조를 인지하여 결합하는 단백질.
2) RNA 결합자리(RNA-binding site) : RNA 결합단백질에서 RNA를 직접 인지하여 결합하는 아미노산 자리.
3) 질량분석(mass spectrometry) : 전체 분자의 질량을 측정한 뒤, 이를 쪼갠 분자 질량을 각각 측정해 분자의 구성을 밝히는 기술.
4) 펩타이드(peptide) : 단백질의 조각으로, 여러 개의 아미노산이 연결돼 이루어진 분자.
5) 유리딘(Uridine) : RNA를 이루는 네 가지 염기 중 하나인 유라실에 탄소 5개를 포함하는 당이 결합한 분자.
연구 추가 설명
논문명
Chemical RNA digestion enables robust RNA-binding site mapping at single amino acid-resolution
저자정보
Jong Woo Bae1,2, S. Chul Kwon1,2, Yongwoo Na1,2, V. Narry Kim1,2,* and Jong-Seo Kim1,2,*
연구이야기
[연구 배경]
RNA 결합단백질은 RNA의 유전자 발현 조절에 핵심적인 기능을 하는데, 이를 연구하기 위해서는 RNA결합자리를 밝히는 것이 매우 중요하다. 기존에는 RNA 조각의 질량이 다양해서 질량 분석 데이터베이스의 크기가 커졌고, RNA결합자리 동정 범위가 매우 작아졌다. 또한, 펩타이드에 결합한 RNA가 질량분석 두 번째 단계 이전에 대부분 조각나기 때문에, RNA의 질량을 이용해 펩타이드 상 RNA결합자리의 위치를 특정하기 어려웠다.
[연구 과정]
불산은 DNA의 인산다이에스터 결합과 펩타이드에 결합한 인산을 분해하는 것으로 알려져 있다. 연구진은 우선 불산이 RNA를 단량체로 완전하게 분해하는지를 초고분해능 액체크로마토그래피를 이용해 확인하였다. 그 다음, 세포에서 RNA-펩타이드 결합체를 추출했다. 불산 처리 후 질량분석을 하여 펩타이드에 결합한 RNA 단량체의 질량을 확인하였다. 펩타이드에 결합해 있던 RNA 분자들은 불산에 의해 극히 단순화되어, 유리딘과 질량분석기 내에서 중간 산물로 형성되는 유라실에 해당하는 분자만이 결합해 있었다. 이 정보를 이용해 세포 전체 RNA 혹은 전령 RNA에 결합한 600 개의 RNA 결합단백질 내 약 2,000 종류의 RNA결합자리를 아미노산 수준 고해상도로 동정할 수 있었다.
추가로 연구진은 이번에 개발한 기법을 이용해 단일 RNA 결합단백질에서 RNA 결합자리를 동정했다. spCas9라는 RNA 결합단백질은 sgRNA와의 결합 구조가 알려져 있다. 동정 결과 기존의 구조 정보와 일치하는 총 84개의 RNA결합자리를 확인했고, 이들은 spCas9의 sgRNA 결합 표면을 따라 분포하였다. 또한 이 중 spCas9의 유전자 편집 기능에 중요한 두 RNA결합자리(Y450, R919)를 규명하였다. 이로부터 RBS-ID의 우수한 정확성뿐만 아니라, 개별 RNA-단백질 결합체 구조와 기능 연구에의 적용 가능성 또한 확인하였다. 이 중 유전자 편집 기능 중요한 RNA결합자리 두 가지 또한 돌연변이에 이은 T7E1 분석을 이용해 규명하였다.
[어려웠던 점]
일반적으로 핵산 분해 효소는 RNA를 단량체로 완전히 분해할 수 있다. 그러나 펩타이드에 결합한 RNA는 구조적인 특성 때문에 완전히 분해할 수 없었다. 연구진은 펩타이드에 결합한 RNA를 단량체로 분해하는 방법을 찾는 데 어려움을 겪었다. 그러던 중 불산이 DNA의 인산다이에스터 결합과 펩타이드에 결합한 인산을 분해한다는 선행 연구를 발견하였다. 이를 펩타이드에 결합한 RNA 분해에 활용하여 난관을 극복할 수 있었다.
[향후 연구계획]
RBS-ID에서 제공하는 정량 정보의 정확성이 높아지게끔 분석 파이프라인을 개선해, 생물학적 조건에 따른 RNA-RNA 결합단백질 상호작용을 아미노산 수준의 해상도로 세밀하게 연구하고자 한다. 또한, UV 이외의 다른 공유결합 형성 방법을 활용해, RBS-ID 기법의 적용 범위를 DNA 결합단백질 연구로 확장시키려 한다.
그림 설명
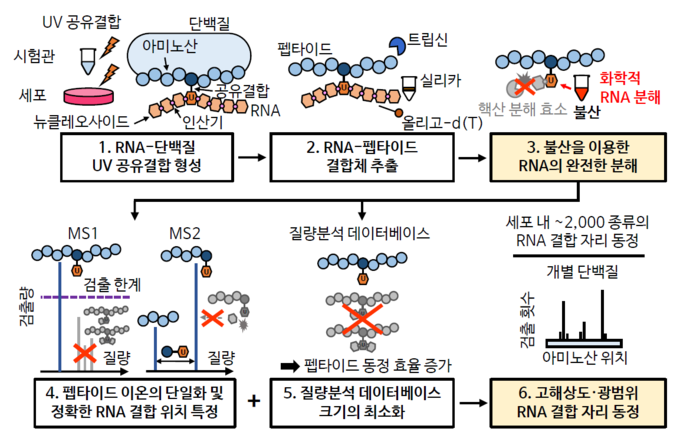
그림 1. 새로 개발한 RBS-ID 실험 기법 모식도.
먼저 UV를 이용해 RNA-RNA 결합단백질 간 공유결합을 형성한다(1).
트립신 효소로 단백질을 분해한 후, RNA-펩타이드 결합체를 추출한다(2).
이후 불산을 이용해 RNA를 완전히 분해한다(3).
이후 분석 과정에서, 펩타이드에 결합한 다양한 RNA 조각들이 불산으로 동일한 분자만 남게 되어, 검출 한계를 넘어서는 RNA-펩타이드 결합체가 많아진다. 또 RNA 조각이 이미 최소화 되었기 때문에 펩타이드를 한 번 더 쪼갤 때(MS2) 더 분해되지 않아, 이를 통해 RNA 결합 위치를 정확히 특정할 수 있다(4).
또, RNA 분자를 한 가지만 고려한 데이터베이스를 사용해, 펩타이드 동정 효율이 증가하게 된다(5). 결과적으로 RBS-ID는 RNA 결합자리를 고해상도·광범위하게 동정할 수 있다.
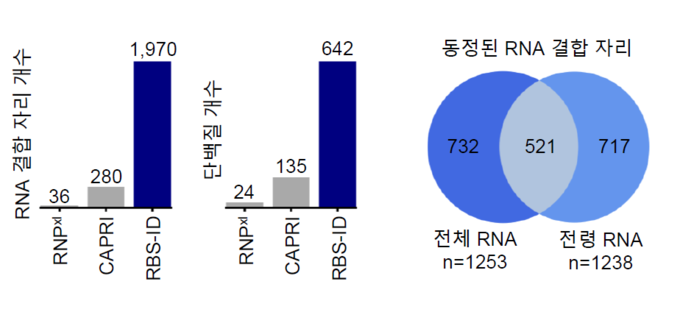
그림 2. RBS-ID의 광범위한 동정 범위 및 기존 기법들과의 비교.
RNPxl과 CAPRI등으로 대표되는 기존의 RNA 결합자리 동정 기법들은 수십-100여 개의 단백질로부터 수십-수백 개의 RNA 결합자리를 동정하는 데 그쳤다.
이에 반해 RBS-ID는 동정 범위를 압도적으로 증가시켜, 세포 내 전체 RNA 및 전령 RNA에 결합하는 약 600 개의 단백질에서 약 2,000 개의 RNA 결합자리를 동정했다.
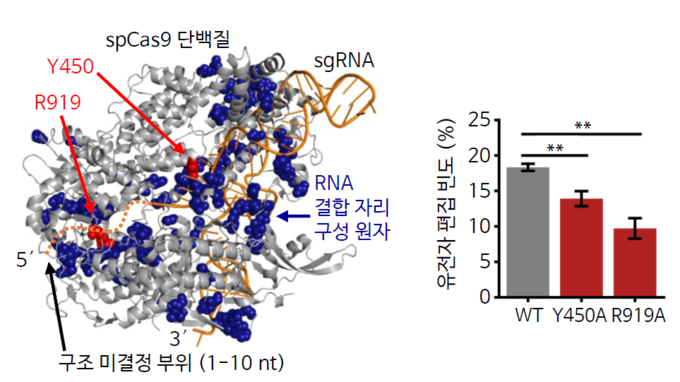
그림 3. spCas9의 RNA 결합자리 동정 및 유전자 편집 기능에 중요한 RNA 결합자리 규명.
이번 연구에서 개발한 기법을 이용, 후속 실험으로 spCas9-sgRNA 결합체에서 84개의 RNA 결합자리를 동정했다. RNA는 결합체 구조에서 spCas9과 sgRNA가 결합하는 표면을 따라 존재한다.
이 중 두 RNA 결합자리(Y450, R919)는 spCas9의 유전자 편집 기능에 중요해, 이 자리들에 돌연변이가 일어나면 spCas9에 의한 유전자 편집 빈도가 감소하는 것으로 드러났다.
그림 4. 연구진 사진.
왼쪽부터 김빛내리 RNA 연구단장(공동교신저자), 배종우 연구원(제1저자), 김종서 책임연구원(공동교신저자). 인물 뒷쪽 기기는 본 프로젝트에 사용한 질량분석기이다(Orbitrap Fusion Lumos, Thermo Fisher Scientific).
연구진 이력사항
[김빛내리 IBS RNA 연구단 단장, 공동교신저자]
1. 인적사항
· 소속 : 기초과학연구원(IBS) RNA 연구단
서울대학교 생명과학부
· 전화 : 02-880-9120
· 이메일 : narrykim@ibs.re.kr / narrykim@snu.ac.kr
2. 학력
· 1992 : 서울대학교 학사 (미생물학과)
· 1994 : 서울대학교 석사 (미생물학과)
· 1998 : 영국 Oxford University 박사 (생화학)
3. 경력사항
· 1999 - 2001 미국 Howard Hughes Medical Institute, University of Pensylvania 박사후연구원
· 2001 - 2004 서울대학교 생명과학인력양성사업단 연구교수
· 2004 - 2008 서울대학교 생명과학부 조교수
· 2007 - 2011 교과부․연구학교 생명과학부 부교수
· 2010 - 2012 교과부․연구재단 지정 국가과학자
· 2010 - 2016 서울대학교 중견석좌교수
· 2012 - 현재 IBS RNA 연구단 단장
· 2013 - 현재 서울대학교 생명과학부 교수
· 2017 - 현재 서울대학교 석좌교수
[김종서 IBS RNA 연구단 책임연구원, 공동교신저자]
1. 인적사항
· 소속 : 기초과학연구원(IBS) RNA 연구단
서울대학교 생명과학부
· 전화 : 02-880-4434
· 이메일 : jongseokim@snu.ac.kr
2. 학력
· 2000 : 서울대학교 학사 (자연과학부 화학전공)
· 2002 : 서울대학교 석사 (화학부 분석화학)
· 2007 : 서울대학교 박사 (화학부 분석화학)
3. 경력사항
· 2007 - 2008 서울대학교 화학부 시간강사
· 2007 - 2008 서울대학교 화학부 박사후연구원
· 2008 - 2013 미국 Pacific Northwest National Laboratory 박사후연구원
· 2013 - 현재 IBS RNA연구단 연구교수/ 서울대학교 책임연구원
[배종우 IBS RNA 연구단 연구원, 제1저자]
1. 인적사항
· 소속 : 기초과학연구원(IBS) RNA 연구단
서울대학교 생명과학부
· 전화 : 02-880-6278
· 이메일 : jwbaebio@snu.ac.kr
2. 학력
· 2018 : 서울대학교 학사 (생명과학부)
· 2018–: 서울대학교 석박사통합과정 재학 (생명과학부)